Kateryna Makova
Originaire d’Ukraine, Kateryna Makova a obtenu son doctorat à la Texas Tech University, où elle a étudié les conséquences génétiques de l’accident de la centrale nucléaire de Tchernobyl. Elle a ensuite effectué ses études postdoctorales à l’université de Chicago, où elle a notamment étudié les différences de taux de mutation entre les mâles et les femelles.
Depuis 2003, elle est professeur au département de biologie de l’université d’État de Pennsylvanie. Son laboratoire mène des recherches en génomique évolutive et médicale. Les sujets d’intérêt actuels comprennent l’évolution des chromosomes sexuels, l’évolution de l’ADN non-B, l’évolution de l’ADN mitochondrial, la variation régionale des taux de mutation et l’obésité infantile. La recherche dans le laboratoire du Dr Makova est hautement interdisciplinaire et collaborative. Le groupe collabore avec des statisticiens, des informaticiens et des biochimistes.
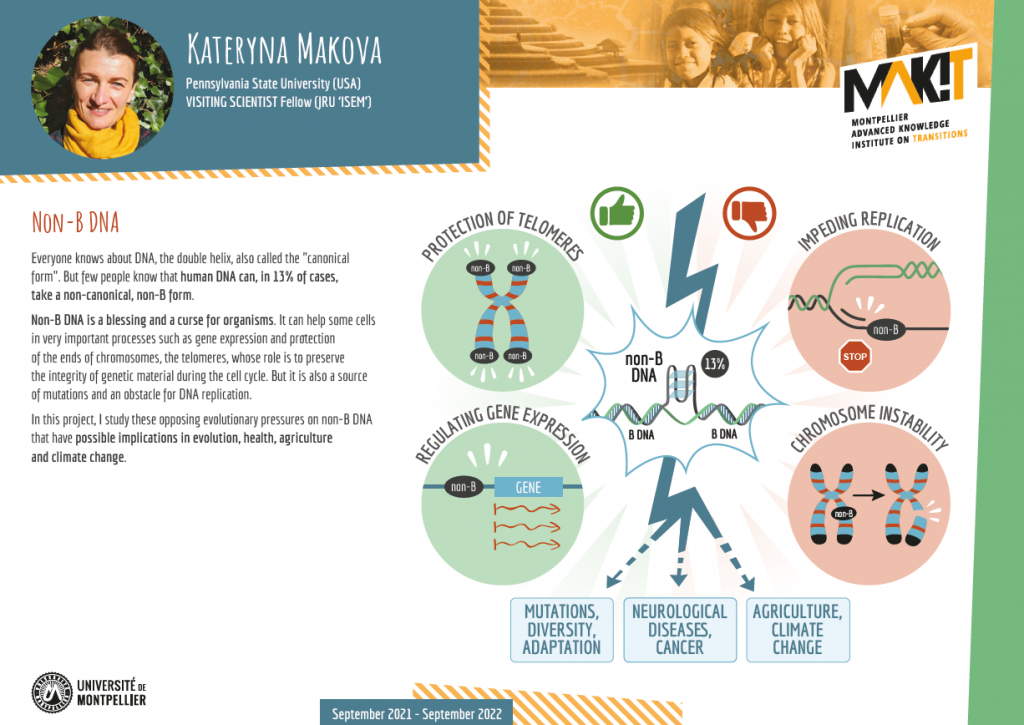
Environ 13 % du génome humain, à certains motifs de séquence, peuvent prendre des conformations différentes de la forme conventionnelle de l’ADN-B décrite par Watson et Crick. Les exemples d’ADN non-B comprennent les quadruplexes G, l’ADN triplex, l’ADN cruciforme et l’ADN-Z. Ces structures remplissent des fonctions importantes dans la cellule, notamment la facilitation du déclenchement des origines de réplication, l’initiation et la répression de la transcription et la protection des télomères. Cependant, l’ADN non-B peut entraver la progression des polymérases pendant la réplication et peut entraîner une instabilité du génome et des taux de mutation élevés.
Dans ce projet, en utilisant des techniques statistiques innovantes du domaine de l’analyse des données fonctionnelles, nous modéliserons les contributions de l’ADN non-B aux variations régionales des taux de mutation. De plus, nous examinerons si l’ADN non-B a joué un rôle important dans les réarrangements du génome au cours de l’évolution des mammifères. Ensuite, nous concevrons de nouveaux tests de sélection statistique, qui tiendront compte des exigences structurelles de l’ADN non-B, et nous identifierons des motifs d’ADN non-B putativement fonctionnels.